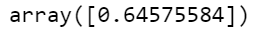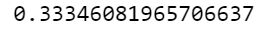러닝스푼즈 수업 정리
< 이전 글 >
https://silvercoding.tistory.com/64
[IRIS 데이터 분석] 1. Python KNN 분류
러닝스푼즈 수업 정리 < 이전 글 > https://silvercoding.tistory.com/63?category=967543 https://silvercoding.tistory.com/62 [boston 데이터 분석] 1. 차원축소 (PCA) 파이썬 예제 러닝스푼즈 수업 정리 라..
silvercoding.tistory.com
데이터 불러오기
이전 글과 동일한 Iris Flower Dataset 을 이용하여 실습을 진행한다.
< Iris Flower Dataset >
https://www.kaggle.com/arshid/iris-flower-dataset
Iris Flower Dataset
Iris flower data set used for multi-class classification.
www.kaggle.com
import pandas as pd
import osos.chdir('../data') # 데이터셋이 있는 본인 폴더 경로iris = pd.read_csv("IRIS.csv")iris.head()
iris['species'].value_counts()
각 종류마다 50개의 데이터가 존재한다.
의사결정나무 사용
train & Test 데이터셋 분리
iris['id'] = range(len(iris))우선 데이터를 구분하기 위해 순서대로 값을 넣어준 id 컬럼을 생성한다.
iris = iris[['id','sepal_length','sepal_width','petal_length','petal_width','species']]id 컬럼이 가장 앞에 오도록 정렬해준다.
train = iris.sample(100,replace=False,random_state=7).reset_index().drop(['index'],axis=1)랜덤으로 100개의 샘플을 추출하여 train 에 저장해 준다.
test = iris.loc[ ~iris['id'].isin(train['id']) ]
test = test.reset_index().drop(['index'],axis=1)train의 id값이 존재하지 않는 iris 데이터들을 test에 넣어준다.
의사결정나무 학습
DecisionTreeClassifier(min_samples_split = n)
---> 특징 : 해석이 쉽고 빠르다.
---> min_samples_split : 의사결정나무에서 최종 노드의 최소 샘플 수
from sklearn.tree import DecisionTreeClassifierdt = DecisionTreeClassifier(min_samples_split = 10)min_samples_split 을 10으로 설정해주어 최종 노드의 샘플수가 10미만이 되지 않도록 조정한다.
dt.fit(train[['sepal_length','sepal_width','petal_length','petal_width']],train['species'])
생성해 놓은 dt 객체로 학습을 시켜준다.
predictions = dt.predict(test[['sepal_length','sepal_width','petal_length','petal_width']])예측값을 prediction에 넣어준다.
test['pred'] = predictions예측값 prediction을 test의 pred 컬럼에 저장한다.
test.head()
(pd.Series(predictions)==test['species']).mean()
예측값과 정답을 비교하여 정확도를 구해보니 0.98이 나왔다.
위의 정확도 측정 방법을 사용하면 신뢰성이 하락할 수 있다. train, test 데이터를 어떻게 나누는지에 따라 결과가 크게 달라질 수도 있기 때문이다. 따라서 cross validation을 이용하여 정확도를 구해볼 수 있다.
from sklearn.model_selection import cross_val_score
import numpy as npdt = DecisionTreeClassifier(min_samples_split = 10)scores = cross_val_score(dt, iris[['sepal_length','sepal_width','petal_length','petal_width']], iris['species'], cv=5, scoring="accuracy")
np.mean(scores)

이번 예시처럼 데이터 수가 적을 경우에는 위와 같이 전체 데이터로 cross validation을 수행하는 것이 신뢰성이 높다. 5 fold cross validation을 수행한 결과 , 정확도가 약 0.97이 나온 것을 볼 수 있다.
의사결정나무 시각화
from sklearn import tree
import matplotlib.pyplot as pltfrom matplotlib.pylab import rcParams
rcParams['figure.figsize'] = 16,10a=tree.plot_tree(dt,feature_names = ['sepal_length','sepal_width','petal_length','petal_width'],impurity=False, max_depth=2, fontsize=10, proportion=True)
plt.show(a)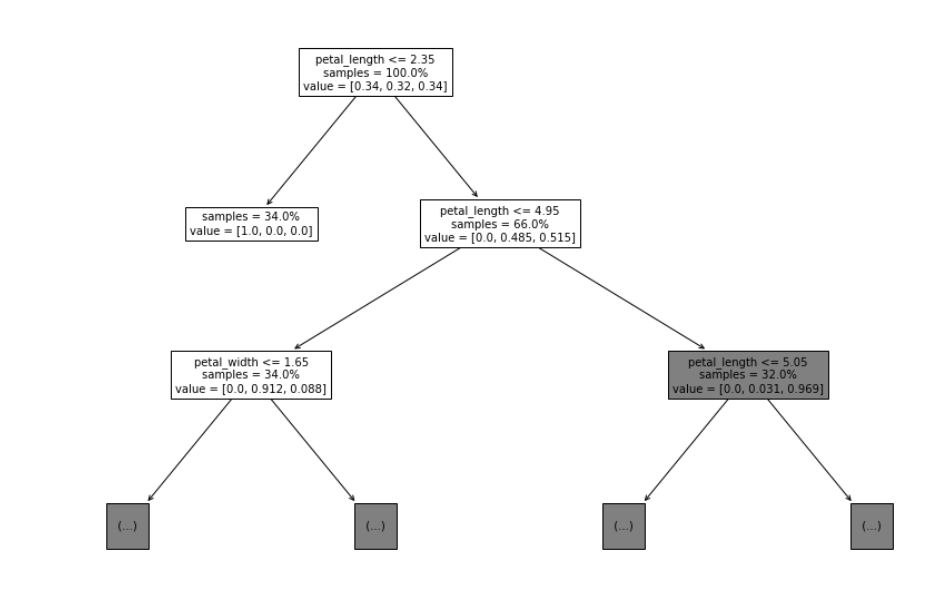
max_depth를 이용하여 깊이를 조절할 수 있다. 2개 이후로는 (...) 으로 생략된 것을 볼 수 있다. 위와 같이 의사결정 나무를 사용하고, 시각화 해보면 해석을 쉽고 간편하게 해낼 수 있다.
'데이터 분석 이론 > 머신러닝' 카테고리의 다른 글
| [Bank Marketing데이터 분석] 2. python 부스팅 Boosting, XGBoost 사용 (0) | 2021.08.23 |
|---|---|
| [Bank Marketing데이터 분석] 1. python 배깅 , 랜덤포레스트 bagging, randomforest (0) | 2021.08.23 |
| [IRIS 데이터 분석] 1. Python KNN 분류 (0) | 2021.08.20 |
| [boston 데이터 분석] 2. PCA, 군집화를 사용한 집값 분석 (0) | 2021.08.19 |
| [boston 데이터 분석] 1. 차원축소 (PCA) 파이썬 예제 (0) | 2021.08.18 |